一 试剂:
CycleTEST PLUS DNA Reagent Kit(BD 公司,340242,40tests)
试剂 A(10ml):含有胰蛋白酶,四氢氯化精胺去污剂,可用于分离实体组织片段,消化 细胞膜及细胞骨架。
试剂 B(8ml):含有胰蛋白酶抑制剂,RNA 酶,柠檬酸盐缓冲液,四氢氯化精胺。用于抑 制胰蛋白酶的作用并消化 RNA。
试剂 C(8ml):含有 PI 染液,四氢氯化精胺,柠檬酸盐缓冲液。PI 作用时的最终浓度至 少有 125ug/ml。
二、培养细胞 DNA 样本的制备
1. 胰酶充分消化,加 PBS 吹打成单个细胞;
2.室温 500×g 离心 5 分钟,吸去上清液,加 1mlPBS 重悬;
3.。 室温 300×g 离心 5 分钟,吸去上清液,加 1mlPBS 重悬;
4.吸去上清液,加入 PBS 重悬细胞,调整细胞浓度到 1.0×106 细胞/ml;
5.取上述浓度的细胞 1ml,室温 300×g 离心 5 分钟,吸去上清液,留下 100ul 左右悬 液;
6.混匀,加入 250ul A 液,轻轻混匀,不要震荡,室温静置 10 分钟;
7. 加入 200ul B 液,轻轻混匀,不要震荡,室温静置 10 分钟;
8.加入 200ul C 液,轻轻混匀,不要震荡,低温(2-8℃)避光静置 10 分钟;
9.用 300 目过滤尼龙网或 50um 尼龙网膜或 35um 细胞过滤器过滤细胞;
10.低温避光放置以待上机检测。建议在加入 C 液后 2 小时内上机,上机前轻轻混匀细胞悬液。
三、流式上机检测:
1、 在浏览框里建文件夹(DNA)和实验组(PI),样本(日期),1 个采集管(样品名称),并重命名。
2、 选择 Cytometer>View Configuration,确保当前的仪器设置包含了合适的参数,本实验需要添加一个 parameter,即 PI,蓝光激发,用 B 接收器接收。
3、 将“浏览框”的采集箭头选中采集管,点击“仪器框”的参数 parameter 页面,删除不必要的荧光参数,仅保留 PI。
4、 所有参数均设为线性,FSC、SSC、PI 选 A 面积信号,另外 PI 还需选择 W 宽度信号。阈值设为 PI,定为 5000。
5、 在 global worksheet 界面建立获取模板:
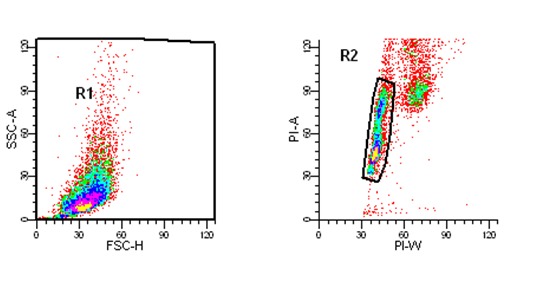
① 一共画三个图:前两个图为散点图,第三个为单参数直方图。 第一个点图,横轴 FSC-A,纵轴 SSC-A。
第二个点图,横轴 PI-W,纵轴 PI-A,。
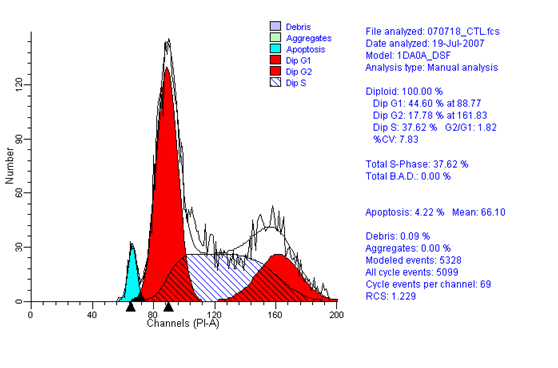
第三个直方图,横轴 PI-A,纵坐标细胞计数。
② 选中第三张图:
⑴ 在100 和200 道数位置设两个间隔门 P1、P2。 并选择“Show Population Hierarchy”。
⑵ 单击右键,选择“Create Statistics View”,
6、上机检测:
①放样品在流式细胞仪上。确认调节机器、获取样本均为低速。
② 确认数据采集箭头指向该管;点击“Acquire”按扭。
③ 看第一张散点图,调整 FSC 和 SSC 电压,使图中细胞群显示在合适位置。
④ 看第二张 PI 散点图(横轴 PI-W,纵轴 PI-A),调节 PI 电压,使得 第一群单核细胞位于纵轴都在100 道数位置。
⑤ 看第三张 PI 直方图,观察单个细胞的 DNAG0-G1 峰也在100 道数位置,若不在100 道位置可以继续调节 PI 电压。
⑥ 调试结束后,在“采集控制框”里选择获取全部 10000 个微粒停止,记录数据。
四、数据导出:
1、 在“D:\BD Export\FCS”目录下,选择“File>New>Folder”命令,新建一个文件夹。
2、 双击打开要导出的实验组,选择“File>Export>Export FCS”命令。
3、 在“Export FCS Files(输出 FCS 文件)”窗口中选择导出文件格式 FSC2.0 或 FSC3.0, 点击 “OK”。
4、 在目录路径的最后,加上您在“驱动盘D:”中新建的文件夹的名字或者用对画框上的“Browser”找到新建文件夹的存储路径。
5、 单击“Save (保存)”。
五、数据分析(ModFIT 软件)
1、 在桌面上双击 ModFIT 图标,打开软件,OK-OK-进入软件主屏;
2、 在 File 下打开数据保存文件夹,文件类型设为 ALL File,选出待分析的文件;
3、 选择参数 Choose parameter of analysis,选 PI-A,双击;
4、 在设门中选择 Defile gate(根据需要,选择是否设门,设几个门)
²Gate 1:X 轴选 FSC,Y 轴选 SSC,OK,然后在图中圈出要分析的细胞;
²Gate 2:X 轴选 PI-W,Y 轴选 PI-A,OK,然后在图中圈出要分析的单个细胞;
²OK 后出现一个直方图。
5、 对这个图进行分析
²自动分析(点击工具栏中的 Auto 键)。
²手动分析
1)单击工具栏中的 Mod 键:选择分析所要的参数(是否有凋亡 Apoptosis, 是否有标准参照 Standard,是否有聚集体),选择有几个 Cycle(二倍体、 四倍体、异倍体),并对每一个循环是否有 S 期和 G2/M 期进行定义-OK;
2)确认每个峰的位置,并将 Range 放置于不同的峰上;
3)单击工具栏中的 Fit 键,软件开始以最小乘法拟合曲线,并进行统计;
6、 Save As 保存分析结果,Print 打印分析结果。
7、 报告中至少应包含以下信息:
⑴ DNA 倍体。G2/G1值.(即G2期是四倍体细胞,而G1期是二倍体细胞,比值应为1.8-2.0之间。若小于1.8,则细胞染色不充分
⑵ 主要 G1 峰的 CV;CV值称为变异系数,一般CV值越小,峰型越好,越尖锐。能控制在5%左右是比较好的结果,一般小于10%就可以认可了。
⑶ S 期比例;
⑷ 必要的简单评价:如细胞数的不足、碎片较多、CV 太高等。